Biochemiczna identyfikacja pałeczek z rodzaju Serratia
Wstępna identyfikacja Serratia sp. na podłożach mikrobiologicznych
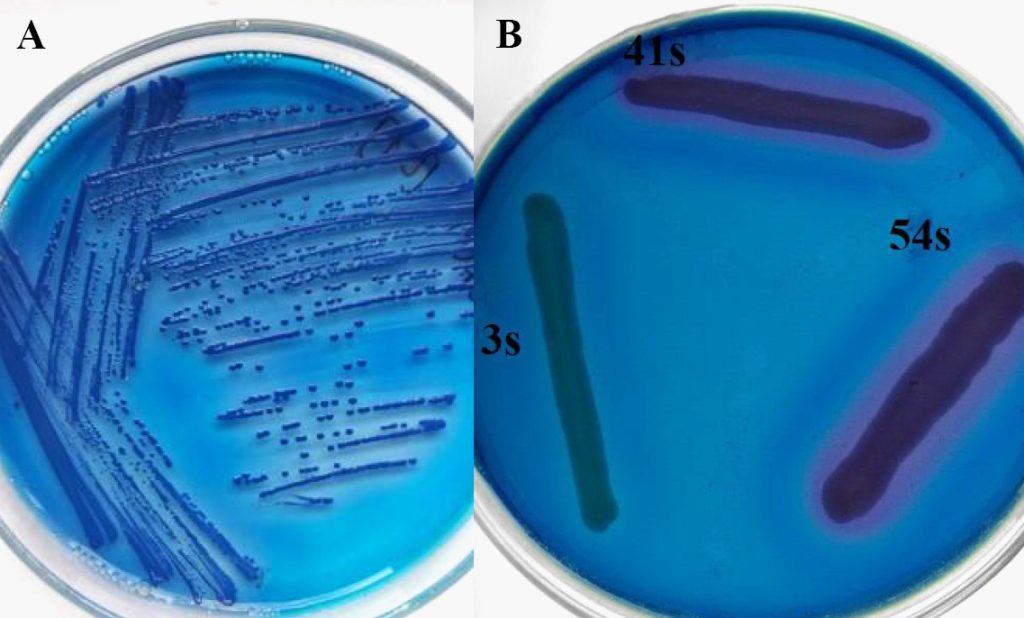
Wstępna identyfikacja bakterii na podłożach mikrobiologicznych pozwala na wyodrębnienie preferowanej grupy spośród tzw. flory towarzyszącej. Dla bakterii Serratia sp. zostało skomponowanych kilka propozycji podłóż umożliwiających izolację i identyfikację tej grupy bakterii z badanych prób pod względem wybranych cech biochemicznych. Podłoża te zaliczane są do grupy podłóż wybiórczych i różnicujących.
W 2006 roku Grimont et. al [3] zaproponował podział podłóż rekomendowanych do izolacji i identyfikacji pałeczek Serratia sp. Przedstawione w tym artykule podłoża to wyłącznie podłoża wybiórcze i są nimi: DTC Agar (deoxyribonuclease-toluidine blue-cephalothin), podłoża DNAse i CT (caprylate-thallous) oraz minimalne podłoże z dodatkiem mezo-erytrytolu. Ich selekcja opiera się na produkcji enzymu deoksyrybonukleazy i oporności na antybiotyki w podłożu DTC agar i podłożu DNAse oraz wykorzystania różnych związków jako źródła węgla [...]
którzy są subskrybentami naszego portalu.
i ciesz się dostępem do bazy merytorycznej wiedzy!